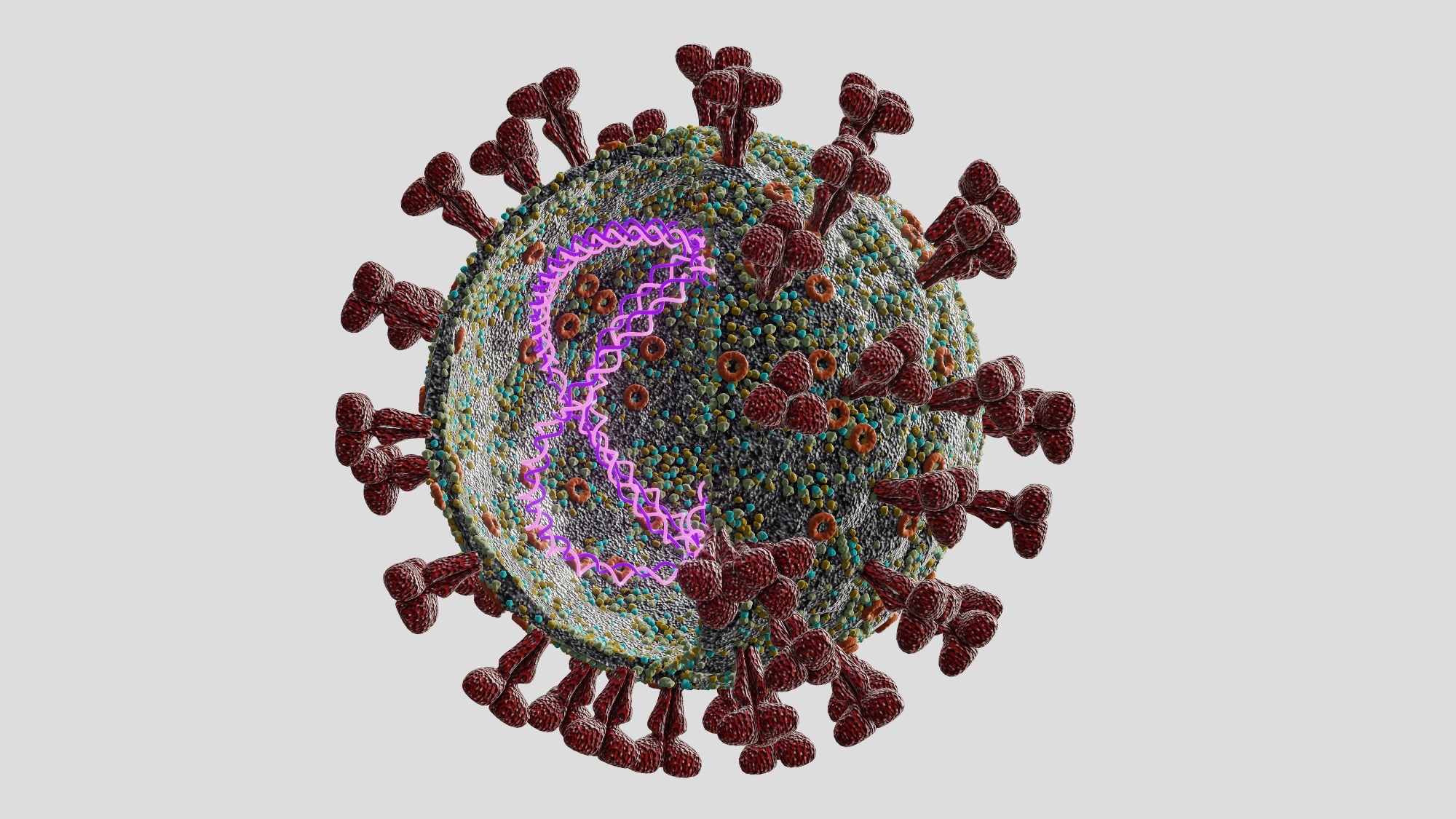
В недавнем исследовании, размещенном на сервере препринтов bioRxiv *, исследователи визуализировали механизм действия и ингибирования неструктурного белка 13 (NSP13) тяжелого острого респираторного синдрома коронавируса 2 (SARS-CoV-2) при высоком пространственно-временном разрешении.
Фон
Из всех 15 NSP SARS-COV-2 NSP13, геликаза рибонуклеиновой кислоты (РНК), имеет решающее значение для его репликации. Однако в настоящее время нет одобренных противовирусных препаратов, нацеленных на nsp13. В отличие от структурных белков SARS-CoV-2, аминокислотная последовательность nsp13 является одной из наиболее консервативных среди многих видов коронавирусов (CoV) (например, ближневосточный респираторный синдром CoV) и вызывающих озабоченность вариантов SARS-CoV-2 (VOC). включая Омикрон. В совокупности это делает nsp13 привлекательной противовирусной мишенью широкого спектра действия с потенциалом для борьбы с будущими вспышками CoV.
Структурные и биохимические исследования показали, что nsp13 представляет собой РНК-хеликазу суперсемейства 1B (SF1B). Он использует механизм дюймовых червей для перемещения вдоль субстратов одноцепочечной (ss) нуклеиновой кислоты (NA), с помощью которого nsp13, вероятно, раскручивает дуплексы NA. Учитывая его небольшой размер шага, методы отдельных молекул не смогли расшифровать скорость, с которой nsp13 перемещается по своему субстрату NA. Такое разрешение могло бы пролить свет на то, как ингибирующие молекулы влияют на их работу.
Об исследовании
В настоящем исследовании исследователи разработали нанопористый пинцет с пикометрическим разрешением (SPRNT) для измерения шагов движения nsp13 SARS-CoV-2 на цепях ДНК. Кроме того, они продемонстрировали, как SPRNT можно использовать для определения механизма действия ингибитора хеликазы. Команда разработала единственную нанопору порина A (MspA) Mycobacterium smegmatis внутри двойного слоя фосфолипидов. Напряжение, приложенное к этой мембране, вызывало протекание ионного тока через нанопору, которая притягивала отрицательно заряженный NA через пору.
Различные основания NA внутри нанопор вызывали уникальные блокировки ионного тока, которые можно было расшифровать в последовательности NA. Хеликаза, связанная с захваченной нитью NA, остановится на краю поры и потянет NA, что приведет к последовательным шагам ионного тока. Команда разделила их на шаги по одному нуклеотиду в субмиллисекундных масштабах времени, чтобы наблюдать за движением геликазы вдоль NA. Параллельно они определили последовательность НК субстрата в геликазе.
Также следует отметить, что SPRNT прикладывал силу, пропорциональную приложенному напряжению, на комплекс фермент/NA, который помогал или сопротивлялся движению nsp13 в зависимости от того, к концу нанопоры был привязан NA. Кроме того, команда наблюдала движение NSP13 вдоль NA в присутствии ингибитора аденозинтрифосфатазы (АТФазы) ATPγS.
Результаты исследования
Исследователи зафиксировали 2413 отдельных событий транслокации и раскручивания NSP13 и 27 641 шаг спирали. Результаты исследования подтвердили, что NSP13 перемещается со скоростью ~1000 нуклеотидов в секунду вдоль одноцепочечной ДНК и раскручивает дуплексы ДНК со скоростью ~100 пар оснований в секунду. Скорость транслокации NSP13 зависела от АТФ, с максимальной скоростью реакции (V max ) в диапазоне от 600 до 3000 с -1 и константой Михаэлиса (K m ) в диапазоне от 100 до 700 мкМ для АТФ, в зависимости от основного контекста последовательности в NSP13. Такие широкие вариации скорости транслокации в разных положениях ДНК указывают на то, что идентичность оснований NA влияет на кинетику транслокации NSP13.
Результаты исследования также показали, что комплекс NSP13-ДНК менее стабилен и легче смещается при приложении силы. Изменение вспомогательной силы от ~24 пикоНьютон (пН) до ~44 пН при насыщении АТФ не вызывало заметного изменения средней скорости перемещения NSP13. Кроме того, это предполагает, что транслокация NSP13 была преимущественно движением, управляемым АТФ-гидролизом.
Авторы также отметили, что шаги раскручивания дуплекса dsDNA (в среднем) были почти в восемь раз длиннее, чем таковые при транслокации ssDNA. Более того, раскручивание dsDNA происходило медленнее, чем транслокация ssDNA, хотя их время пребывания коррелировало. Аналогичный эффект наблюдался в другом исследовании, посвященном изучению PcrA геликазы SF1A с использованием SPRNT. Интересно, что РНК-зависимая РНК-полимераза SARS-CoV-2 (RdRp) и NSP13 образуют комплекс со скоростью примерно 170 нуклеотидов в секунду при 37 °C, аналогичный тому, что наблюдалось при скорости раскручивания NSP13 с использованием SPRNT.
Кроме того, авторы отметили, что ATPγS препятствует действию NSP13 через несколько различных кинетических процессов. Однако доминирующий механизм зависел от применения вспомогательной силы. Хотя ATPγS не является жизнеспособным лекарственным средством для NSP13, он продемонстрировал силу SPRNT при изучении механизмов ингибирования хеликазы. Было выявлено три метода ингибирования NSP13:
и) снижение его процессивности,
ii) предотвращение присоединения его доменов 1A и 2A после связывания нуклеотидов, и
iii) замедление гидролиза ATPγS в большей степени по сравнению с АТФ.
Выводы
В целом исследование показало, что SPRNT является ценным и мощным инструментом для изучения роли NSP13 в комплексе репликации и транскрипции SARS-CoV-2 (RTC). Метод SPRNT также показал превосходную способность облегчить изучение кинетики транслокации NSP13 или любой хеликазы, даже без дуплекса. Кроме того, эксперименты SPRNT могут облегчить изучение NSP13 на нативных последовательностях SARS-CoV-2, чтобы пролить свет на конкретные элементы последовательности высокоструктурированного генома SARS-CoV-2 и их роль в регуляции NSP13.
*Важное замечание
bioRxiv публикует предварительные научные отчеты, которые не рецензируются экспертами и, следовательно, не должны рассматриваться как окончательные, направляющие клиническую практику/поведение, связанное со здоровьем, или рассматриваться как установленная информация.






















Оставить Комментарий
Вы должны войти, чтобы оставить комментарий.