
За последние несколько десятилетий было разработано очень мало новых антибиотиков, в основном потому, что современные методы скрининга потенциальных лекарств непомерно дороги и требуют много времени. Одной из многообещающих новых стратегий является использование вычислительных моделей, которые предлагают потенциально более быстрый и дешевый способ идентификации новых лекарств.
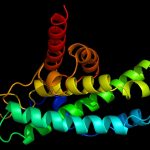
Новое исследование Массачусетского технологического института раскрывает потенциал и ограничения одного из таких вычислительных подходов. Используя белковые структуры, созданные программой искусственного интеллекта под названием AlphaFold, исследователи изучили, могут ли существующие модели точно предсказывать взаимодействия между бактериальными белками и антибактериальными соединениями. Если это так, то исследователи могут начать использовать этот тип моделирования для крупномасштабного скрининга новых соединений, нацеленных на ранее нецелевые белки. Это позволит разработать антибиотики с беспрецедентным механизмом действия, что является важной задачей для преодоления кризиса устойчивости к антибиотикам.
Однако исследователи во главе с Джеймсом Коллинзом, профессором медицинской инженерии и науки Термеера в Институте медицинской инженерии и науки Массачусетского технологического института (IMES) и Департаменте биологической инженерии, обнаружили, что эти существующие модели не подходят для этой цели. На самом деле их предсказания сработали немногим лучше, чем случайность.
Прорывы, такие как AlphaFold, расширяют возможности поиска лекарств in silico, но эти разработки необходимо сочетать с дополнительными достижениями в других аспектах моделирования, которые являются частью усилий по открытию лекарств. Наше исследование говорит как о текущих возможностях, так и о текущих ограничениях вычислительных платформ для открытия лекарств».
Джеймс Коллинз , профессор медицинской инженерии и науки в Термеере Института медицинской инженерии и науки Массачусетского технологического института (IMES) и факультета биологической инженерии.
В своем новом исследовании исследователи смогли улучшить производительность этих типов моделей, известных как моделирование молекулярной стыковки, путем применения методов машинного обучения для уточнения результатов. Однако, по словам исследователей, потребуются дополнительные улучшения, чтобы в полной мере использовать белковые структуры, предоставляемые AlphaFold.
Коллинз — старший автор исследования, опубликованного сегодня в журнале Molecular Systems Biology . Постдоки Массачусетского технологического института Феликс Вонг и Арти Кришнан являются ведущими авторами статьи.
Молекулярные взаимодействия
Новое исследование является частью недавно начатого лабораторией Коллинза проекта Antibiotics-AI Project, целью которого является использование искусственного интеллекта для открытия и разработки новых антибиотиков.
AlphaFold, программное обеспечение для искусственного интеллекта, разработанное DeepMind и Google, точно предсказало структуру белков по их аминокислотным последовательностям. Эта технология вызвала волнение среди исследователей, ищущих новые антибиотики, которые надеются, что смогут использовать структуры AlphaFold для поиска лекарств, связывающихся со специфическими бактериальными белками.
Чтобы проверить осуществимость этой стратегии, Коллинз и его студенты решили изучить взаимодействие 296 основных белков кишечной палочки с 218 антибактериальными соединениями, включая антибиотики, такие как тетрациклины.
Исследователи проанализировали, как эти соединения взаимодействуют с белками кишечной палочки , используя моделирование молекулярной стыковки, которое предсказывает, насколько сильно две молекулы будут связываться друг с другом, исходя из их формы и физических свойств.

California Gold Nutrition, Lactobif, Cran-Max, пробиотики, 25 млрд КОЕ, 30 растительных капсул
Этот тип моделирования успешно использовался в исследованиях, в которых проводится скрининг большого количества соединений в сравнении с одним белком-мишенью, чтобы идентифицировать соединения, которые лучше всего связываются. Но в этом случае, когда исследователи пытались сравнить множество соединений со многими потенциальными мишенями, предсказания оказались гораздо менее точными.
Сравнивая прогнозы, полученные с помощью модели, с фактическими взаимодействиями для 12 основных белков, полученными в ходе лабораторных экспериментов, исследователи обнаружили, что частота ложноположительных результатов модели была аналогична истинным положительным результатам. Это говорит о том, что модель не смогла последовательно определить истинные взаимодействия между существующими лекарствами и их мишенями.
Используя измерение, часто используемое для оценки вычислительных моделей, известное как auROC, исследователи также обнаружили низкую производительность. «Используя эти стандартные симуляции молекулярной стыковки, мы получили значение auROC примерно 0,5, что в основном говорит о том, что вы делаете не лучше, чем если бы вы угадывали случайным образом», — говорит Коллинз.
Исследователи получили аналогичные результаты, когда они использовали этот подход к моделированию со структурами белка, которые были определены экспериментально, вместо структур, предсказанных AlphaFold.
«Похоже, AlphaFold работает примерно так же хорошо, как и экспериментально определенные структуры, но нам нужно лучше работать с моделями молекулярного докинга, если мы собираемся эффективно и широко использовать AlphaFold при открытии лекарств», — говорит Коллинз.
Лучшие прогнозы
Одна из возможных причин плохой работы модели заключается в том, что белковые структуры, подаваемые в модель, статичны, в то время как в биологических системах белки гибки и часто меняют свою конфигурацию.
Чтобы попытаться повысить вероятность успеха своего подхода к моделированию, исследователи прогнали прогнозы с помощью четырех дополнительных моделей машинного обучения. Эти модели обучаются на данных, которые описывают, как белки и другие молекулы взаимодействуют друг с другом, что позволяет им включать больше информации в прогнозы.
«Модели машинного обучения изучают не только формы, но и химические и физические свойства известных взаимодействий, а затем используют эту информацию для переоценки прогнозов стыковки», — говорит Вонг. «Мы обнаружили, что если вы будете фильтровать взаимодействия с помощью этих дополнительных моделей, вы можете получить более высокое соотношение истинных положительных результатов к ложным положительным результатам».
Тем не менее, исследователи говорят, что необходимо дополнительное усовершенствование, прежде чем этот тип моделирования можно будет использовать для успешной идентификации новых лекарств. Один из способов сделать это — обучить модели большему количеству данных, включая биофизические и биохимические свойства белков и их различные конформации, а также то, как эти свойства влияют на их связывание с потенциальными лекарственными соединениями.
По словам Коллинза, благодаря дальнейшим достижениям ученые смогут использовать возможности белковых структур, созданных искусственным интеллектом, для открытия не только новых антибиотиков, но и лекарств для лечения различных заболеваний, включая рак. «Мы с оптимизмом смотрим на то, что с улучшением подходов к моделированию и расширением вычислительной мощности эти методы будут становиться все более важными при открытии лекарств», — говорит он. «Однако нам предстоит пройти долгий путь, чтобы полностью раскрыть потенциал открытия лекарств in silico».
Исследование финансировалось Фондом Джеймса С. Макдоннелла, Швейцарским национальным научным фондом, Национальным институтом аллергии и инфекционных заболеваний, Национальными институтами здравоохранения и Институтом Броуда Массачусетского технологического института и Гарварда. Проект Antibiotics-AI поддерживается Audacious Project, Flu Lab, Sea Grape Foundation и Wyss Foundation.




















Оставить Комментарий
Вы должны войти, чтобы оставить комментарий.